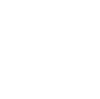Интерпретация исследования
В результате исследования вы получите информацию о патогенных вариантах, ассоциированных с исследованными заболеваниями.
В заключении будут отражены наследуемые или вновь возникшие (de novo) однонуклеотидные замены, инсерции и делеции размером до 10 пар нуклеотидов в кодирующих регионах (экзонах) и сайтах сплайсинга. К техническим ограничениям метода относятся:
- крупные инсерции и делеции (более 10 пар нуклеотидов);
- мутации в интронных областях;
- вариации количества повторов;
- мутации в генах, у которых имеется псевдоген в составе генома;
-мутации в сложных участках генома (например, GC-богатые участки).
В заключение не включаются синонимичные варианты, варианты в интронных областях генов. Кроме того, в заключение не включаются распространенные варианты, которые ранее не были описаны как патогенные.
Одновременное обследование обоих супругов позволяет сократить диагностический поиск. Результат будет направлен на электронную почту, указанную при оформлении заказа.
Для получения клинической интерпретации заключения необходима консультация врача- генетика.
Список генов:
AAAS, AARS1, AARS2, AASS, ABCA1, ABCA3, ABCA4, ABCB11, ABCB4, ABCC2, ABCC6, ABCC8, ABCG5, ABCG8, ACAD8, ACAD9, ACADM, ACADS, ACADSB, ACADVL, ACAT1, ACE, ACP5, ACTA2, ACTC1, ACVRL1,
ACY1, ADA, ADA2, ADAMTS13, ADAR, ADGRG1, ADGRV1, ADPRS, ADSL, AGA, AGK, AGL, AGT, AGXT, AHI1, AIPL1, AIRE, AKR1C2, ALAD, ALDH5A1, ALDH6A1, ALDH7A1, ALDOB, ALG1, ALG11, ALG12, ALG3,
ALG6, ALG8, ALMS1, ALOX12B, ALOXE3, ALPL, AMPD1, AMT, ANO10, ANO5, AP4B1, AP4M1, AP4S1, AP5Z1, APC, APOB, APOC2, APOE, APRT, APTX, AQP2, ARFGEF2, ARSA, ARSB, ASL, ASPA, ASPH, ASPM,
ASS1, ASXL1, ATM, ATP2A1, ATP6V1B1, ATP7B, ATP8B1, ATR, AUH, AXIN2, B3GAT3, B3GLCT, B4GALT7, BAG3, BAP1, BARD1, BBS1, BBS10, BBS12, BBS2, BBS5, BBS7, BCHE, BCKDHA, BCKDHB, BCS1L, BLM,
BMPR1A, BMPR1B, BRAT1, BRCA1, BRCA2, BRIP1, BSND, BTD, C12ORF57, C19ORF12, C2, C8B, CABP2, CACNA1C, CACNA1H, CACNA1S, CACNA2D4, CAPN3, CARS2, CASP8, CASQ2, CASR, CBS, CC2D2A,
CCDC103, CCDC39, CCDC65, CD36, CD3G, CDH1, CDH2, CDH23, CDH3, CDHR1, CDKN1B, CENPJ, CEP152, CEP290, CEP83, CETP, CFAP410, CFH, CFI, CFTR, CHAT, CHEK2, CHRNA3, CHRNA4, CHRNB1, CHRND, CHRNE, CHRNG, CLCN1, CLCN2, CLCNKB, CLDN14, CLDN16, CLN3, CLN5, CLN6, CLN8, CLPB, CLRN1, CNGA3, CNGB1, CNGB3, COL11A2, COL18A1, COL1A2, COL3A1, COL4A3, COL4A4, COL6A1, COL6A3, COL7A1, COQ2, COQ4, COQ8A, COX20, CP, CPA6, CPLANE1, CPS1, CPT1A, CPT2, CR2, CRADD, CRB1, CSPP1, CSTB, CTC1, CTH, CTLA4, CTNS, CTU2, CUBN, CUL7, CWC27, CYB5R3, CYBA, CYP11B1, CYP11B2, CYP1B1, CYP21A2, CYP24A1, CYP27A1, CYP27B1, CYP3A4, CYP7B1, DARS2, DBH, DBT, DCDC2, DCHS1, DCLRE1C, DDC, DDHD2, DDX11, DES, DHCR7, DHODH, DHTKD1, DIS3L2, DLD, DMD, DNAAF11, DNAAF3, DNAAF4, DNAH11, DNAH5, DNAI1, DNAJB2, DNM2, DNMT1, DOCK6, DOCK8, DOK7, DONSON, DPYD, DPYS, DRC1, DSC2, DSG1, DSG2, DSP, DUOX2, DUOXA2, DYNC2H1, DYNC2I2, DYSF, EARS2, ECEL1, ECHS1, ECM1, EFNB1, EGFR, EIF2B2, EIF2B5, EIF3F, ELN, ELP1, ENG, ENO3, ENPP1, EPCAM, EPM2A, ERCC2, ERCC3, ERCC4, ERCC5, ERCC6, ERCC6L2, ESCO2, ETFDH, ETHE1, EVC, EXOSC3, EYS, F12, F13A1, F2, F5, F7, F9, FAH, FAN1, FANCA, FANCC, FANCD2, FANCG, FANCI, FANCL, FBN1, FBN2, FBP1, FBXL4, FECH, FGFR3, FGG, FH, FIG4, FITM2, FKBP10, FKBP14, FKTN, FLCN, FLG, FLNC, FMO3, FOLR1, FOXE3, FOXRED1, FRAS1, FSHR, FTCD, FUT2, G6PC1, GAA, GALC, GALK1, GALNS, GALNT3, GALT, GBA, GBE1, GCDH, GCH1, GCK, GDAP1, GFER, GFM1, GFPT1, GGCX, GHSR, GINS1, GJB2, GLA, GLB1, GLDC, GLE1, GMPPB, GNB1, GNE, GNPTAB, GNPTG, GNRHR, GP1BA, GP6, GP9, GPAA1, GPC3, GPIHBP1, GPR179, GRHPR, GRM6, GRN, GUCY2D, GYG1, GYS2, H6PD, HADHA, HARS1, HAX1, HBB, HCFC1, HEXA, HEXB, HFE, HGD, HGSNAT, HINT1, HMGCL, HMGCS2, HNF1A, HNF4A, HOGA1, HPD, HPS1, HPS3, HRAS, HSD17B3, HSD17B4, IDUA, IFT140, IFT27, IGF1R, IGHMBP2, IL12RB1, IL1RAPL1, IL2RA, IL36RN, ILDR1, IMPG2, INS, INVS, IQCE,IRAK4, ITGA2B, ITGB3, ITPA, IVD, KATNB1, KCNE1, KCNH2, KCNJ1, KCNJ11, KCNQ1, KCTD7, KDSR, KIAA0586, KIAA0753, KIF7, KIT, KIZ, KLHL41, KPTN, KYNU, LAMA2, LAMB3, LAMC3, LARP7, LARS1, LARS2, LDLR, LIPA, LIPT1, LMBRD1, LMNA, LOXHD1, LPIN1, LPL, LRMDA, LRP5, LSS, LTBP2, LTBP3, LZTR1, MAN2B1, MAOA, MAP1B, MASP2, MAX, MC2R, MC4R, MCCC1, MCCC2, MCEE, MCOLN1, MED12, MED17, MEFV, MEN1, MET, MFN2, MFRP, MFSD8, MITF, MKKS, MKS1, MLH1, MMAA, MMAB, MMACHC, MME, MMP1, MMUT, MNS1, MOGS, MPI, MPL, MPO, MPV17, MPZL2, MRE11, MRPS34, MSH2, MSH3, MSH6, MTFMT, MTHFD1, MTHFR, MTO1, MTR, MTRR, MUSK, MUTYH, MVK, MYBPC3, MYH11, MYH7, MYH8, MYL2, MYL3, MYLK, MYMK, MYO15A, MYO3A, MYO6, MYO7A, NADSYN1, NAGA, NAGLU, NBAS, NBN, NCF1, NCF4, NDE1, NDUFA6, NDUFB3, NEB, NEK8, NF1, NF2, NHLRC2, NIPAL4, NMNAT1, NOD2, NPC1, NPHP3, NPHP4, NPHS1, NPHS2, NPPA, NR2E3, NTHL1, NTRK1, NUBPL, NUP93, OCA2, ODAD1, OPA1, OPCML, OPLAH, ORC6, OTC, OTOA, OTOF, OTOG, OTOGL, P2RY12, PADI3, PAH, PALB2, PANK2, PAPSS2, PARS2, PCCA, PCCB, PCK1, PCSK9, PDE6B, PDE6H, PDGFRA, PDZD7, PEX1, PEX10, PEX12, PEX5, PEX6, PEX7, PGM1, PHKB, PHOX2B, PHYH, PIBF1, PIGG, PIGL, PIGN, PIGQ, PIGT, PIGV, PISD, PKHD1, PKLR, PKP2, PLA2G6, PLCE1, PLEKHG5, PLG, PLOD1, PLOD2, PMM2, PMP22, PMS2, PNKP, PNPLA6, PNPO, PNPT1, POC1B, POLD1, POLE, POLG, POLR1C, POLR3A, POLR3B, POMC, POMGNT1, POMT1, POP1, POR, POT1, POU4F1, PPA2, PPARG, PPP1R21, PPT1, PRDX1, PRF1, PRKAG2, PRKAR1A, PRKN, PRMT7, PROC, PRODH, PROM1, PROP1, PROS1, PRSS56, PTCH1, PTEN, PTH, PTH1R, PTPN23, PTPRQ, PTS, PUS3, PYCR1, PYGL, PYGM, PYROXD1, RAB23, RAB27A, RAC1, RAC2, RAD50, RAD51C, RAG1, RAPSN, RARS1, RARS2, RB1, RBM20, RBM8A, RBP3, RCBTB1, RDH12, RDH5, RECQL4, RET, RHO, RIPK4, RMND1, RMRP, RNASEH2B, RNU4ATAC, RPE65, RSPH1, RTEL1, RUNX1, RYR1, RYR2, SBDS, SCN2A, SCN4A, SCN5A, SCO2, SCYL1, SDHA, SDHAF2, SDHB, SDHC, SDHD, SDR9C7, SEC23B, SECISBP2, SELENON, SERPINA1, SERPINB7, SERPINB8, SERPINC1, SERPINF1, SFTPB, SGCA, SGCE, SGSH, SI, SIL1, SLC12A3, SLC13A5, SLC17A5, SLC22A12, SLC22A5, SLC24A1, SLC25A13, SLC26A2, SLC26A3, SLC26A4, SLC2A2, SLC2A9, SLC34A1, SLC34A3, SLC37A4, SLC39A13, SLC39A8, SLC3A1, SLC45A2, SLC4A1, SLC52A3, SLC5A2, SLC5A5, SLC6A19, SLC6A3, SLC7A7, SLC7A9, SLC9A3R1, SLCO1B1, SLURP1, SLX4, SMAD3, SMAD4, SMARCA2, SMARCA4, SMARCAL1, SMARCB1, SMARCE1, SMPD1, SOD1, SPAG1, SPG11, SPG7, SPINK1, SPINK5, SPTB, SQSTM1, SRD5A3, STAC3, STAR, STAT3, STIL, STK11, STRC, STUB1, SUFU, SUMF1, SUOX, SURF1, SYNE1, SYNE4, TAT, TBC1D24, TDP1, TECPR2, TELO2, TERT, TF, TG, TGFBR1, TGFBR2, TGM1, TGM5, THOC6, THRA, TIA1, TK2, TMC1, TMEM126B, TMEM127, TMEM199, TMEM216, TMEM43, TMEM67, TMPRSS15, TMPRSS3, TNFRSF13B, TNFRSF1A, TNNC1, TNNI3, TNNT1, TNNT2, TOE1, TOR1A, TP53, TPK1, TPM1, TPO, TPP1, TRAPPC11, TRDN, TREX1, TRIM32, TRIOBP, TRIP13, TRIT1, TRMU, TRNT1, TRPM1, TRPM6, TRPV6, TSC1, TSC2, TSEN54, TSFM, TSHR, TTC12, TTC19, TTC21B, TTC8, TTLL5, TTN, TTPA, TTR, TUBB3, TUBB8, TUBGCP2, TUSC3, TWNK, TYMP, TYR, UBA5, UGT1A1, UNC13D, UPB1, USH1C, USH2A, VARS2, VDR, VHL, VKORC1, VPS13A, VPS13B, VPS37A, VWA1, VWF, WARS2, WDR35, WDR62, WFS1, WIPI2, WNT10A, WRN, WT1, WWOX, XDH, XPA, XRCC2, XRCC4, ZBTB24, ZFYVE26, ZMPSTE24
по результатам исследования ДНК методом клинического секвенирования
Пациент:
Пол: Женский Дата рождения:
Направительный диагноз:
Вид исследования: 1950 Скрининг на наследственные заболевания "Расширенный" Дата забора материала:. Дата исследования:
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Варианты, которые могут быть вероятной причиной заболевания приоритезированы по проприетарному алгоритму с учетом рекомендаций ACMG, наличию в базах данных, популяционных частот и других критериев.
На основании проведенной приоритезации и фенотипа пациента, описанного в представленных документах варианты сгруппированы по степени вероятности их патогенности для пациента. В группах варианты расположены в порядке снижения приоритетности.
Варианты, не имеющие признаков патогенности, либо имеющие некоторые признаки патогенности, но, несоответствующие фенотипу, описанному в сопроводительных документах, могут быть не включены в заключение.
Подробно с описанием исследования можно ознакомиться в приложении к заключению.
ВНИМАНИЕ! Варианты, обнаруженные в результате исследования, не являются установленным диагнозом, а могут быть использованы в совокупности с данными других лабораторных и инструментальных методов только врачом генетиком.
Для уточнения значимости обнаруженных вариантов, в том числе с учетом клинической картины пациента необходима консультация врача-генетика.
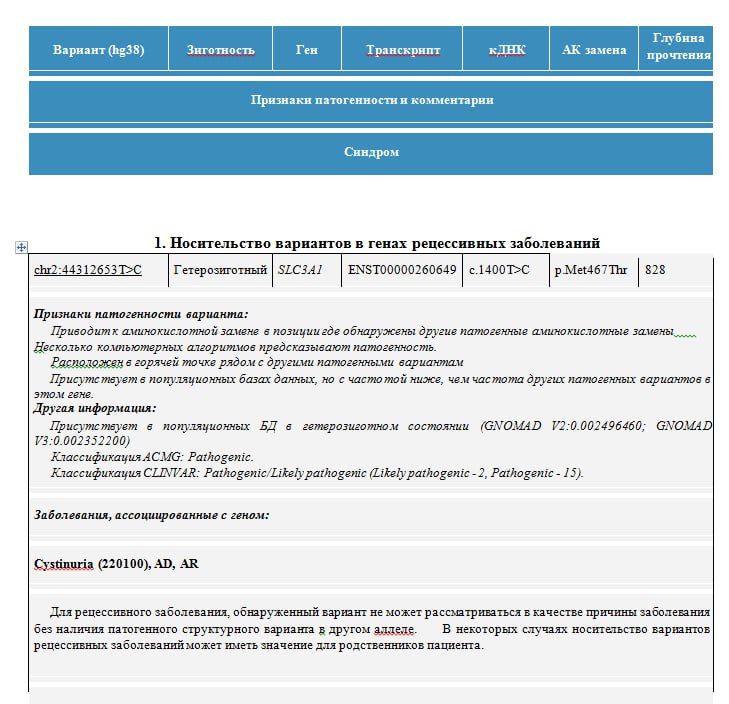
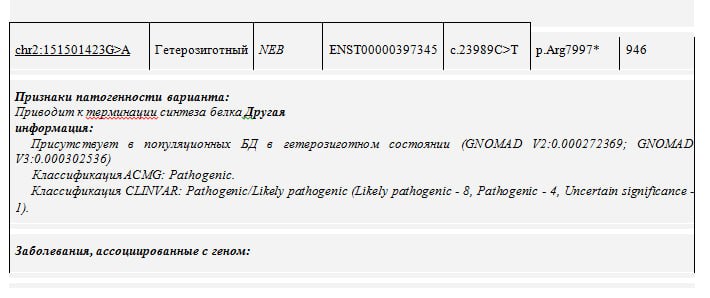
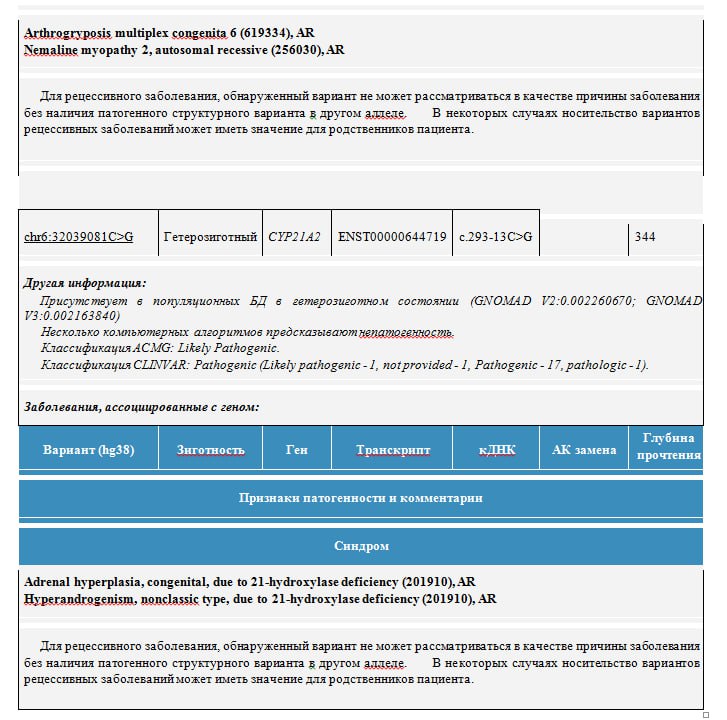
1. Носительство вариантов в генах рецессивных заболеваний